L’Off Topic che vi proponiamo oggi tratta un argomento un po’ borderline per essere considerato Off Topic ma abbiamo comunque preferito metterlo qua perché non tratta la tecnologia consumer che trattiamo ogni giorno.
In questi giorni, tramite amici, ho sentito parlare di Folding @ Home, un progetto sviluppato e gestito dal laboratorio Pande alla Stanford University, sotto la direzione del professor Vijay Pande, ed è condiviso da varie istituzioni scientifiche e laboratori di ricerca in tutto il mondo.

Fondamentalmente il progetto è nato per aiutare i medici nella lotta ad alcune malattie importanti come Malattia di Alzheimer, Malattia di Huntington, Cancro, Osteogenesi Imperfetta e nella lotta ai Virus.
In misura minore Folding @ Home cerca anche di prevedere la struttura finale di una proteina e di determinare come altre molecole vadano ad interagire con essa: questa è un’attività molto utile nella Progettazione di Farmaci.
Il progetto è salito alla ribalta in questo periodo perché è in prima linea per la lotta al COVID-19.
Cosa è Folding@Home?
Come riporta Wikipedia, Folding@home (talvolta abbreviato come FAH o F@h) è un progetto che utilizza il calcolo distribuito per simulare e studiare diversi fenomeni, quali il ripiegamento delle proteine, la progettazione di farmaci e altri tipi di dinamiche molecolari. Il progetto usa la potenza di calcolo inutilizzata di migliaia di PC di proprietà di volontari, che hanno deciso di installare e di eseguire un apposito software sul proprio computer. Il suo scopo principale è quello di determinare i meccanismi di ripiegamento delle proteine, che è il processo mediante il quale le proteine raggiungono la loro struttura tridimensionale finale, e di esaminare le cause che portano ad errati ripiegamenti delle stesse.
Cosa fa in due parole?
In pratica, una volta registrati, riceverete dei micro-pacchetti di modelli da sviluppare che verranno elaborati dalla vostra CPU/GPU in modo tale che ogni computer connesso ai server di Folding @ Home esegua questi “calcoli” per poi reinviare i risultati elaborati che verranno messi insieme.
I volontari potranno poi tenere traccia dei loro contributi sul sito web di Folding @ Home sotto forma di un punteggio basato sul lavoro totale svolto, il che rende la partecipazione dei volontari competitiva ed incoraggia il coinvolgimento a lungo termine.
Il Software
Il software di Folding @ Home è disponibile per Linux, Mac e Windows, ma per l’elenco completo vi rimandiamo a questo link.

Il software per l’utente è composto da tre componenti:
– Work Unit
– Core
– Client
Può essere eseguito sia in background mentre l’utente svolge altre operazioni che quando il computer è in disuso:
“Mentre continui con le tue attività quotidiane, il tuo computer lavorerà per aiutarci a trovare cure per malattie come cancro, SLA, Parkinson, Huntington, Influenza e molti altri.“
In ogni caso è possibile settare il livello di risorse dedicate a Folding @ Home su tre livelli (light, medium e full) a libera scelta per l’utente.
Man mano che il PC dell’utente elabora un pacchetto dati, accumula dei punti: questi punti sono parte di una sorta di “sfida” tra tutti gli utenti che partecipano al progetto.

La figura sopra è il web control di Folding @ Home. In alto a destra viene mostrato il numero di punti accumulati mentre in basso e possibile trovare i dati sul “progetto” al quale si sta contribuendo: vengono mostrate la Work Unit e alcune informazioni sul progetto. Running sottolinea che stiamo condividendo le nostre risorse.
Poco più in basso troviamo la configurazione rapida sul tipo di potenza di calcolo (Power) da condividere e quando farlo: se mentre lavoriamo o solo se quando il pc è inutilizzato.
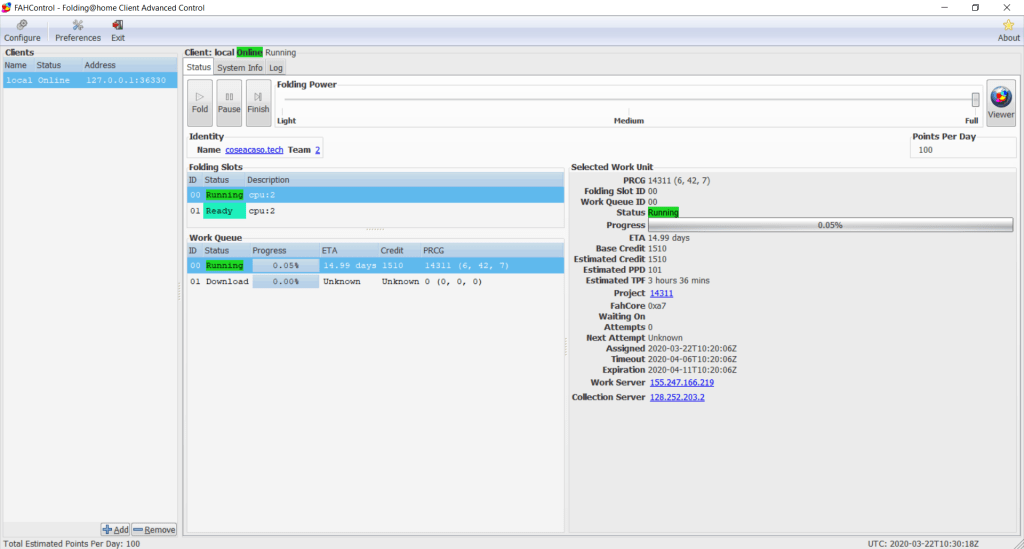
Quella mostrato sopra è il pannello dell’Advanced Control. Si tratta della versione avanzata e completa (e con grafica meno accattivante) di quanto troviamo nel Web Control: in questa console troviamo informazioni complete su tutto ed è possibile modificare in maniera “fine” la configurazione di quanto si voglia offrire.
Per non effettuare un copia-incolla da Wikipedia, vi rimandiamo ancora una volta alla pagina sull’argomento per approfondire circa la funzionalità delle varie componenti.
La lotta al COVID-19
Abbiamo ormai tutti imparato a conoscere il Coronavirus e tutto ciò che ha comportato e sta comportando a livello globale. Sappiamo che non è ancora stato effettivamente trovato un vaccino per curare la COVID-19 ma che il ceppo è stato isolato e la ricerca è nel pieno del proprio sviluppo.
A questo punto, entra in campo Folding @ Home.
Lo scorso 27 Febbraio, è arrivata la richiesta d’aiuto di Folding @ Home dove veniva annunciato l’inizio della lotta contro COVID-19/2019-nCOV.
“Abbiamo bisogno del tuo aiuto! Folding @ home si unisce ai ricercatori di tutto il mondo che lavorano per comprendere meglio il Coronavirus 2019 (2019-nCoV) per accelerare lo sforzo scientifico aperto per sviluppare nuove terapie salvavita. Scaricando Folding @ Home , puoi donare le tue risorse computazionali inutilizzate al consorzio Folding @ home , dove i ricercatori che lavorano per far progredire la nostra comprensione delle strutture di potenziali bersagli farmacologici per 2019-nCoV che potrebbero aiutare nella progettazione di nuove terapie. I dati che ci aiutate a generare saranno rapidamente e apertamente diffusi nell’ambito di una collaborazione scientifica aperta di più laboratori in tutto il mondo, offrendo ai ricercatori nuovi strumenti che potrebbero sbloccare nuove opportunità per lo sviluppo di farmaci salvavita.”
Questa illustrazione, creata presso i Centers for Disease Control and Prevention (CDC), rivela la morfologia ultrastrutturale esibita dai Coronavirus.
“2019-nCoV è un cugino vicino al coronavirus SARS (SARS-CoV) e agisce in modo simile. Per entrambi i coronavirus, il primo passo dell’infezione si verifica nei polmoni, quando una proteina sulla superficie del virus si lega a una proteina del recettore su una cellula polmonare. Questa proteina virale è chiamata proteina spike , rappresentata in rosso nell’immagine seguente, e il recettore è noto come ACE2. Un anticorpo terapeutico è un tipo di proteina che può bloccare il legame della proteina virale al suo recettore, impedendo quindi al virus di infettare la cellula polmonare. È già stato sviluppato un anticorpo terapeutico per SARS-CoV, ma per sviluppare anticorpi terapeutici o piccole molecole per 2019-nCoV, gli scienziati devono comprendere meglio la struttura della proteina virale virale e come si lega al recettore ACE2 umano necessario per l’ingresso virale nelle cellule umane.“
“Le proteine non sono stagnanti: si muovono, si piegano e si aprono per assumere numerose forme. Dobbiamo studiare non solo una forma della proteina virale virale, ma tutti i modi in cui la proteina si muove e si piega in forme alternative al fine di comprendere meglio come interagisce con il recettore ACE2, in modo da poter progettare un anticorpo. Esistono strutture a bassa risoluzione della proteina di picco SARS-CoV e conosciamo le mutazioni che differiscono tra SARS-CoV e 2019-nCoV. Alla luce di queste informazioni, siamo in una posizione unica per aiutare a modellare la struttura della proteina del picco 2019-nCoV e identificare i siti che possono essere presi di mira da un anticorpo terapeutico. Siamo in grado di costruire modelli computazionali per raggiungere questo obiettivo, ma richiede molta potenza di calcolo.“
“Una proteina del 2019-nCoV, una proteasi codificata dall’RNA virale, è già stata cristallizzata . Sebbene la proteina di picco di interesse del 2019-nCoV non sia stata ancora risolta (ma sembra legata ad ACE2), il nostro obiettivo è utilizzare la struttura omologa della proteina di picco della SARS-CoV per identificare i target terapeutici degli anticorpi.“
Proprio per questi motivi, è importantissimo avere molti computer che lavorano per lo stesso obiettivo: sviluppare un rimedio terapeutico il più rapidamente possibile.
Scaricando Folding @ Home e selezionando la funzione per contribuire a “Any Disease” (qualsiasi malattia), si fornisce il potere computazionale necessario per affrontare questi problemi.
Trovate il tutto sul sito: www.foldingathome.org
